大豆食心虫成虫的虫卵会附着于大豆表面,孵化的幼虫会啃食大豆,对大豆产量和品质造成严重影响。这种现象在我国各大豆产区普遍发生,如发现和预防得不及时会使得大豆产量严重下降。在大豆病害的检测上,人工可视化调查作为实践中最基本的直接方法,至今仍在使用。然而,这种方法需要相关植物表型和植物病理学的专业知识;另一种常见的植物病害检测技术可以称为生物分子法,但生物分子技术需要详细的取样和复杂的处理方法,与人工调查方法相比,这些方法更具专业性和周期性。这两种技术具有基本性、有效性,但总是需要手动去检测,导致复杂的工作和较大的劳动量。
近年来,深度学习技术在植物病害分类中的成功应用,为大豆病害的研究提供了新思路,该技术通过将卷积网络与高光谱结合,能够对目标进行有效分类。高光谱技术可视为光谱学的一部分,它可以从多个光谱带中获取光谱信息,一些有效波段对病害引起的大豆细微变化具有很高的敏感性,从而可以区分不同的病害类型。卷积网络是一种特殊的深度学习技术,在图像处理和提取特征方面具有突出的能力。
1实验部分
1.1样本制备
测试集中所需要食心虫大豆样本由专业农业机构提供,将只成虫放入大豆中使其于大豆上产卵,5d后采集附着虫卵的大豆,10d后采集附着食心虫幼虫的大豆,30d后采集被啃食的大豆。分别对正常的大豆以及上述三种大豆拍摄高光谱图像,每类样本数量为20。
1.2高光谱成像系统
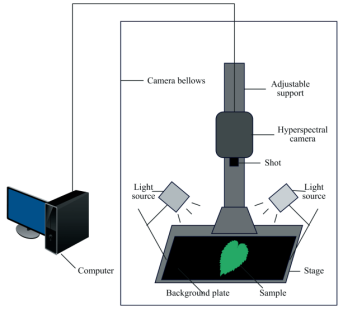
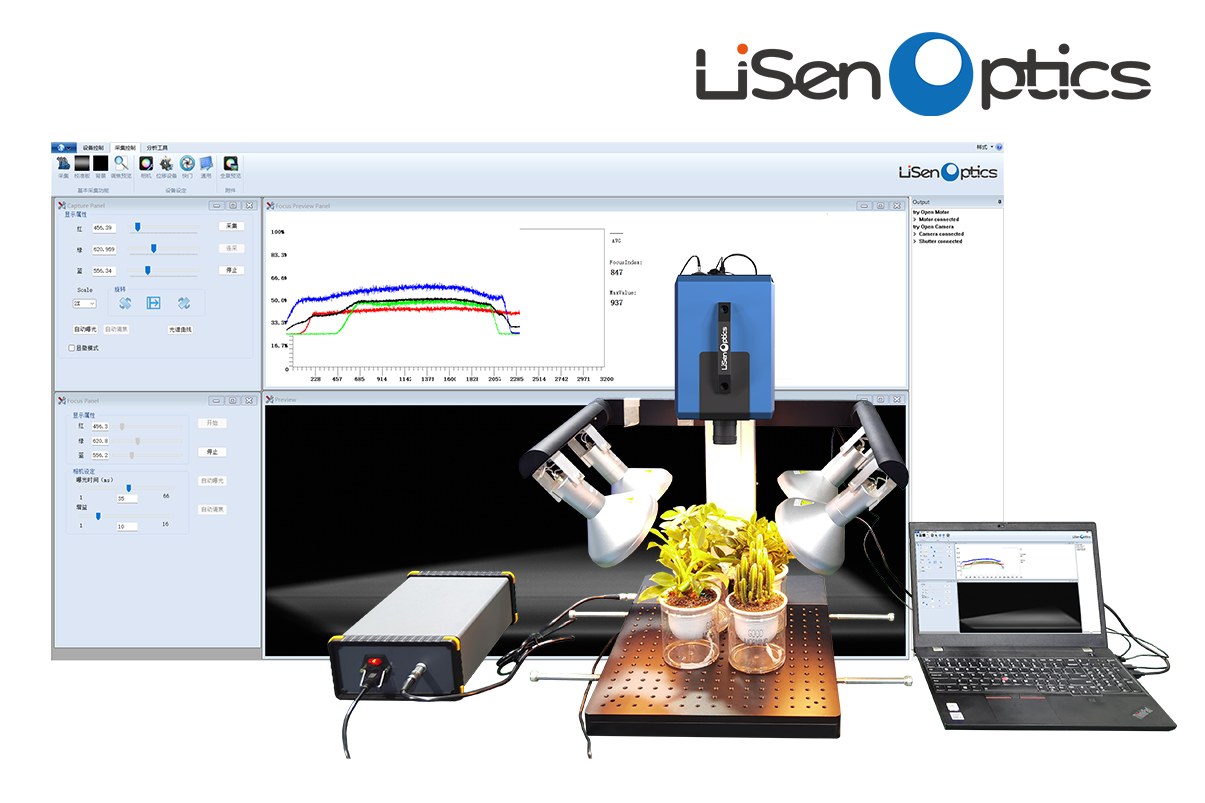
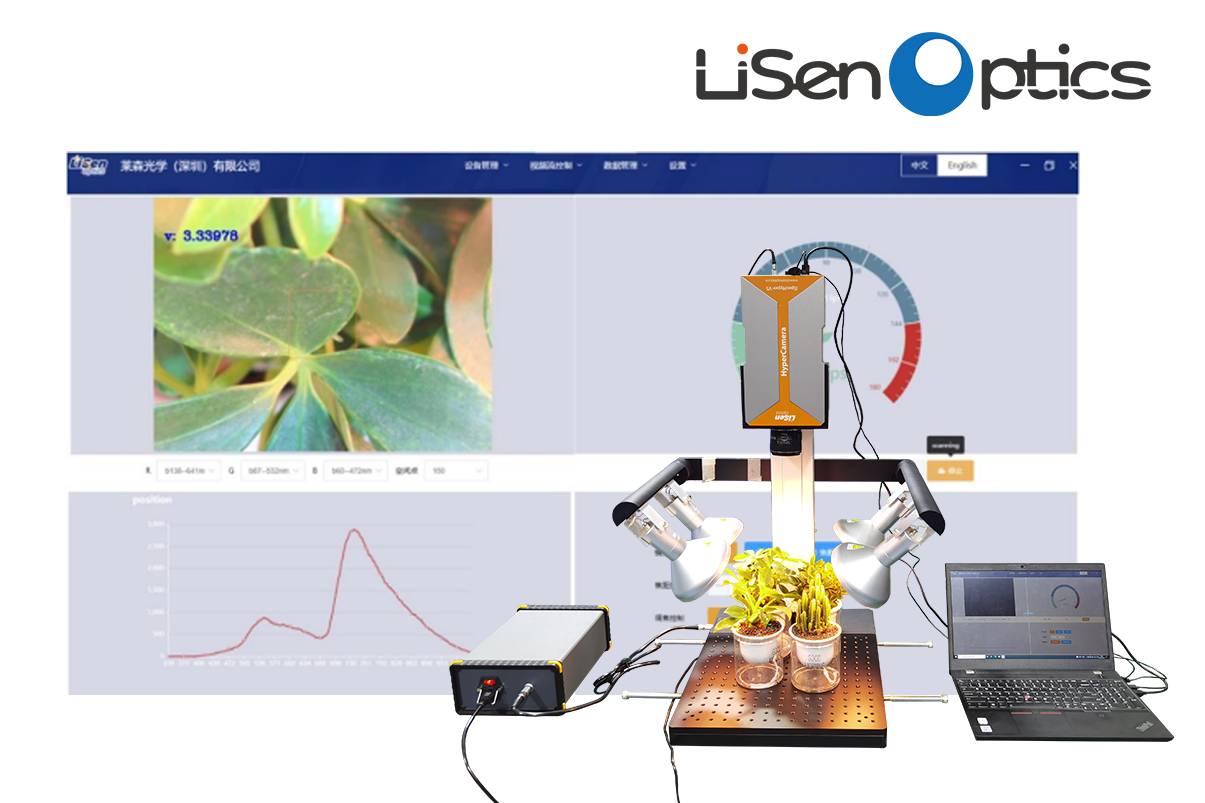
由软件进行图像采集,高光谱成像系统的组成部分有电控平移台,4盏功率为150w的卤素灯,CCD相机,一台计算机五个部分,采集的图像包含256个光谱波段。它们的光谱范围为383.70~1032.70nm,整个采集过程在暗箱中完成,避免了环境光的影响,如图1所示。

图1系统结构图
1.3高光谱图像采集
采集图像时,曝光时间为18ms,平台的移动速度设置为1.50cm.s-1,卤素灯与平台之间的夹角设置为50°。先采集白板图像W和暗背景图像B,然后对大豆样本进行图像采集,得到256个波段的高光谱图像。采集到的大豆样本合成的RGB图如图2所示
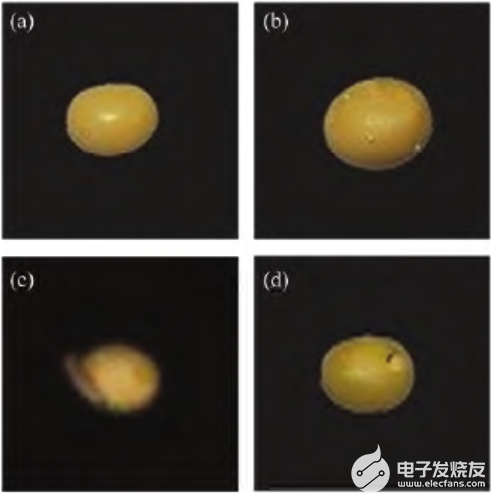
图2大豆样本高光谱图像
1.4数据预处理
采集图像过程中为了缓解可能出现的漫反射,样本不均匀,基线漂移等问题,对采集图像进行了Savitzky-Golay滤波处理,针对光照不均匀的问题,使用了黑白校正的预处理方法。高光谱图像的波段数量太多,许多波段的数据是冗余的;为了避免这些冗余数据造成的影响,使用了主成分分析法进行降维处理,选取前30个高光谱波段作为特征波段。以样本为中心截取了50*50像素的正方形区域,使得单个高光谱样本数据的大小为50*50*30。
1.5图像检索模型原理
3D-R-D模型的内容主要包括一个用于提取特征的3D卷积网络,一个用于帮助网络产生有效特征的DCH损失函数。其中网络模型为3D-Resnet18,该预训练模型的原始数据集包括Kinetics-700(K)和MomentsinTime(M)两个数据集,前者一共700个类,每个类包括超过600个来自You-Tube的人类动作视频,后者是包括100万个视频的数据集,使用这样的预训练模型,可以得到较好的初始化参数。预训练模型并不能直接使用,在网络结构的改变上,本工作仅仅去除最后的分类层,并添加一个从高维映射到低维的hash层,这样不仅使用了模型中的初始化参数,还能利用网络训练来进行降维。在损失函数的设计上,为得到效果优良的相似度特征,文献中经常使用成对损失函数来更新这些特征提取算法的可学习参数。专家学者通过设计合理的损失函数,使得每一对输入样本如果相似就让它们的特征相互靠近,不相似的样本对特征距离相互远离,充分利用了样本标签之间的相似性;部分学者对输入图像的监督信息进行编码,对输出特征进行正则化,以逼近所需的离散值,该方法设计了一个阈值m,当不相似对的特征距离大于m时不提供损失贡献。部分学者设计了一个基于柯西分布的成对损失,它对汉明距离大于给定汉明半径阈值的相似图像对造成了显著的惩罚,也充分利用了标签信息。将中的损失函数与交叉熵函数进行结合,使得最后的特征信息包含分类损失和标签之间的相似度信息。通过引入一个哈达码矩阵,改进交叉熵损失,通过添加分类中心,增强了分类损失的特征信息。在特征距离的计算上,由于最后的特征都是低维的二进制码,所以使用汉明距离进行衡量,并且由于维度较低,不同样本之间特征距离的计算速度极快。
2结果与讨论
2.1模型使用与分析
如上所述。本工作使用预训练模型,并将分类层改为hash层,损失函数使用的是DCH,训练集为CAVE,iCVL和NUS,测试集为采集的大豆样本,训练时用于微调以适应应用场景的训练集则和文献中描述的一样,就是使用光谱数据集CAVE,iCVL和NUS作为数据集。CAVE是一个包含32个场景的数据集,iCVL高光谱数据集由计算机视觉会议收集,包含农村,城市,植物,公园,室内这些场景,NUS数据集则包含了一些普通场景和水果,而上述的四种类别的大豆数据则作为分类实验中的测试集和检索集。利用图像检索进行分类的步骤如下所示:
(1)利用训练集训练得到一个网络模型,这个模型能够对输入的同一类高光谱样本输出相似的二进制特征。
(2)从大豆样本的每一类中随机取出10个样本组成检索集,对检索集提取特征并储存到文件A中,剩下的大豆样本作为测试集。
(3)vwin 新采集样本的分类:将测试集中每一个样本依次提取特征,并用汉明距离与文件A中的数据进行相似度匹配排序,从检索集取出的排名前5个样本中相同标签最多的即为这个测试样本的类别。
(4)实验得到的准确率即为正确的测试个数占总测试样本数量的比重,重复上述步骤,反复实验取平均值。在最近的大豆食心虫分类研究中,文献使用了小样本分类,采用了MN,MAML,3D-RN的方法,也解决了高光谱图像数据样本不足的问题,它的实验结果如表1所示。
表1不同分类模型在4-way5-shot情况下的检测结果
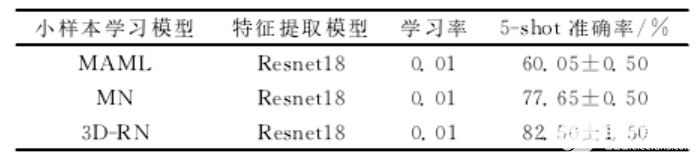
由此发现,使用Resnet18的网络结构,具有良好的效果,为了研究图像检索进行分类的有效性,也为了能和文献进行对比,通过使用不同的预模型,结合不同损失函数,设计了如表2实验。
表2不同损失函数下的检索性能
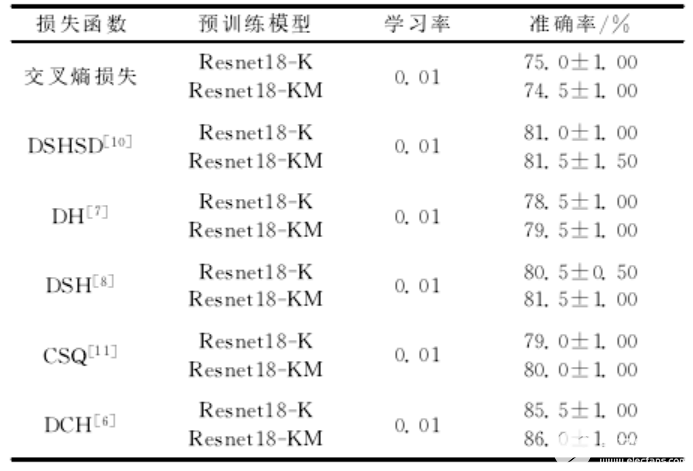
在表2中,Resnet18-K表示使用了Kinetics-700(K)为数据集的预训练模型,Resnet18-KM,表示该预训练模型还使用了MomentsinTime(M)数据集,从实验结果中发现,尽管使用了更多的数据,本实验在性能上,有所提升但是比较细微;和原来的交叉熵损失函数相比,DH使用成对损失提高了5%的分类准确率,在使用M阈值之后,DSH又有所提高,DSHSD尽管结合分类损失和成对损失,但是性能和DSH几乎没有区别,由此可见,单纯的分类损失并不适用于本次实验,CSQ引入了中心损失,在改善交叉熵后,确实提高了5%左右的准确率,和DSH差不多,而DCH在使用柯西分布后,达到了86%的准确率,相比于之前效果最好的3D-RN实验,提高的准确率。
3结论
近年来,使用高光谱成像对农业病虫害检测已经应用地十分广泛,但是样本数量少的问题仍然需要解决,本文通过采集不同时期的样本,利用样本之间的相似度信息,构建了一种基于图像检索的分类方法,在模型上,从视频检索中得到启发,利用大量数据训练的3D预训练网络,获得了较好的初始化参数,利用3DCNN,使得不同波段间数据的相似性能够被利用起来,通过对不同损失函数的比较,DCH利用柯西分布,能较好地提取到样本之间地相似信息,它的准确率达到了86.0±1.00,从而解决了实际问题,这是一种新颖的高光谱检测方法,为高光谱检测的相关研究提供了一种新的思路。
审核编辑 黄昊宇
-
光谱检测
+关注
关注
0文章
12浏览量
6641 -
高光谱
+关注
关注
0文章
330浏览量
9934
发布评论请先 登录
相关推荐
做病虫害监测,要如何选择合适的地物光谱仪厂家
如何使用高光谱成像技术进行作物健康监测?

友思特分享 | 清晰光谱空间:全自动可调波长系统的高光谱成像优势




 基于图像检索的大豆食心虫虫害高光谱检测
基于图像检索的大豆食心虫虫害高光谱检测













评论